多伦多大学、DeepMind等开发BioReason使AI像生物学家一样推理
编辑 | 萝卜皮
听到基因组、蛋白质组等词汇,大家首先想到的是什么?庞杂的数据?得出的结论解释性差?
虽然 DNA 基础模型拥有强大的序列表征能力,但是难以进行多步推理,并且缺乏内在透明、生物学直观的解释。
在这里,来自多伦多大学(University of Toronto)、Vector Institute、DeepMind 的研究团队推出了 BioReason,这是一种开创性的架构,它首次将 DNA 基础模型(Evo2)与 LLM (Qwen3)深度集成。
这种新颖的连接方式使 LLM 能够直接处理基因组信息并将其作为基本输入进行推理,从而形成一种全新的多模态生物学理解形式。
该研究以「BioReason:Incentivizing Multimodal Biological Reasoning within a DNA-LLM Model」为题,于 2025 年 5 月 29 日发布在 arXiv 预印平台。

展开全文
论文链接:
具体来讲,BioReason 运行在两个主要输入流上:(i) 一个或多个基因组序列,以及 (ii) 文本查询。
DNA 基础模型将每个输入的 DNA 序列转换为语境化的嵌入,而大型语言模型则充当主要推理引擎和文本生成器。这种集成的关键在于 DNA 嵌入块的准备,其中基因组信息通过将 DNA 嵌入与用户查询的嵌入以及特殊标记(例如 <dna_start> 和 <dna_end>)堆叠在一起,集成到 LLM 的输入中。
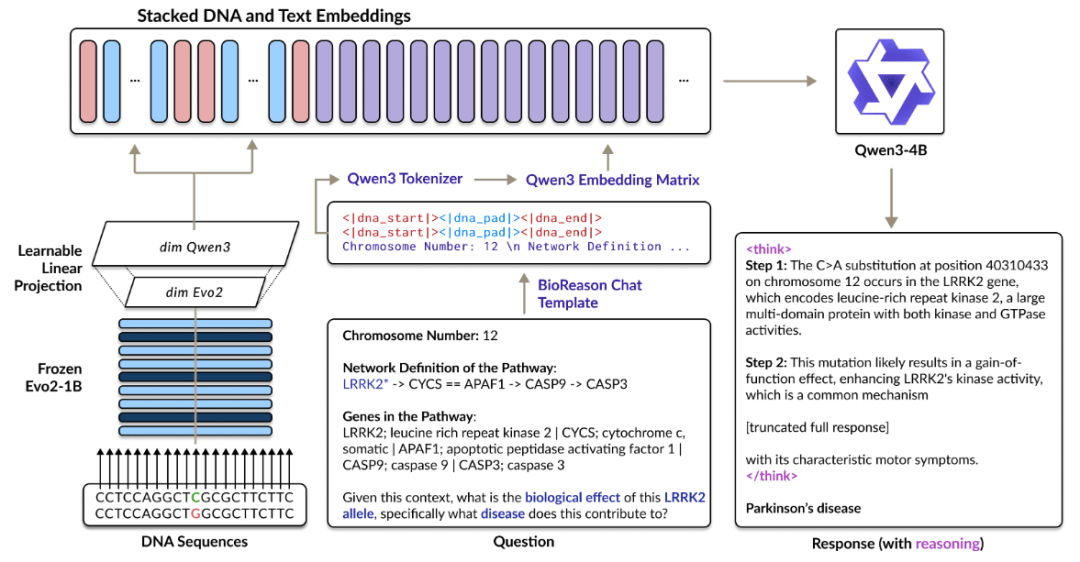
图示:BioReason 的架构。(来源:论文)
性能评估
研究人员精心挑选了三个全面的数据集用于训练和评估:一个基于 KEGG 的全新生物推理数据集(1,449 条条目),用于阐明遗传变异与疾病表型之间的机制联系;一个针对编码序列的变异效应预测数据集(50,083 条条目);一个用于编码非 SNV 的数据集(36,088 条条目)。
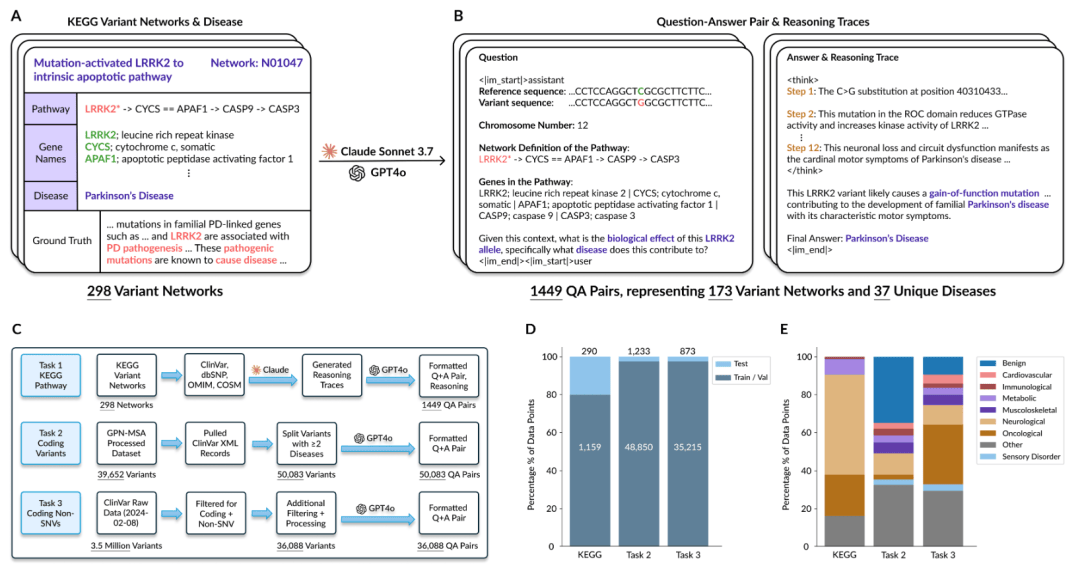
图示:BioReason 数据集管理和组成。(来源:论文)
KEGG 数据集使用标准化符号表示分子网络,包括激活、抑制和调控相互作用;而 VEP 数据集则侧重于跨不同基因组变异的致病/良性分类和疾病表型预测。
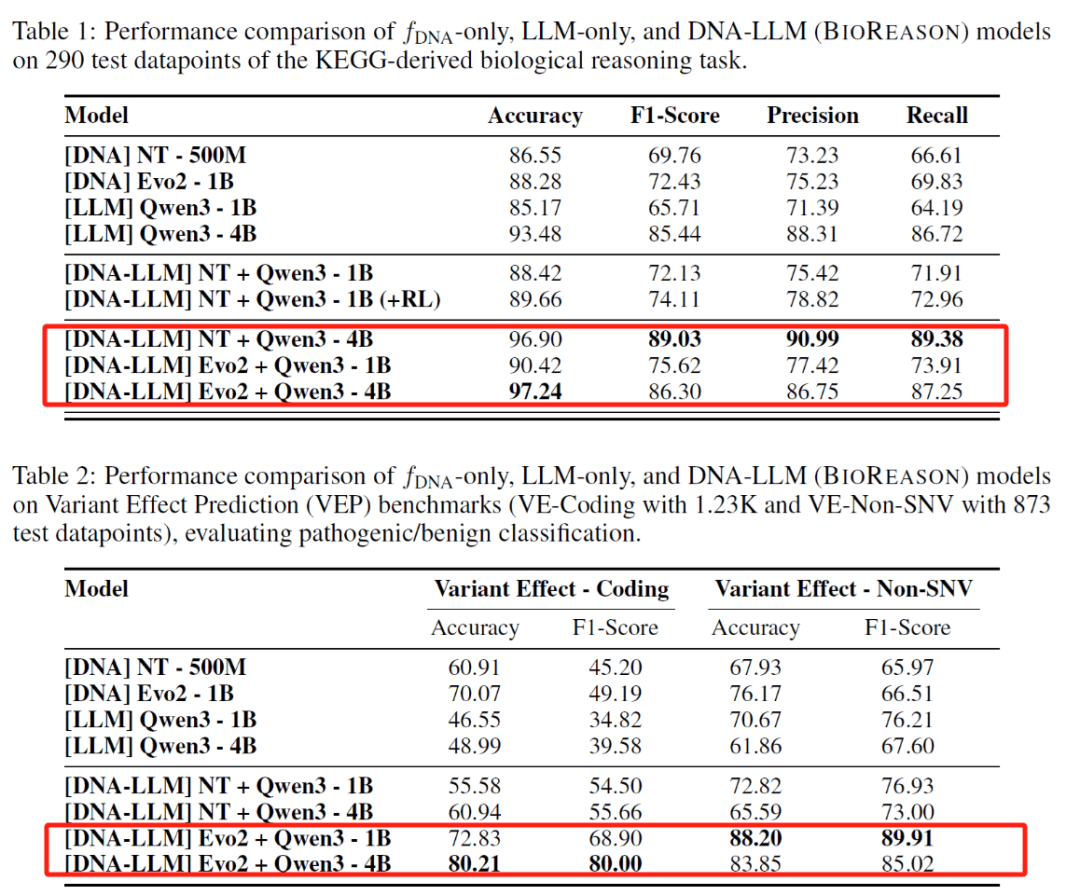
图示:基于三个数据集对 BioReason 的性能进行评估。(来源:论文)
在 KEGG 衍生的推理基准测试中,Evo2+Qwen3-4B 模型的准确率达到 97.24%,F1 得分达到 86.30%。在变异效应预测 (VEP) 任务中,Evo2+Qwen3-4B 模型的编码变异准确率达到 80.21%,非 SNV 分类准确率达到 88.20%,在所有任务中均显著优于仅基于 DNA 和 LLM 的基线模型。
案例研究
为了测试 BioReason 的可解释推理能力,研究人员提出一个假想案例,假设通路上下文为「肌动蛋白(单体)//PFN1*//肌动蛋白(丝状)」,则询问 PFN1 等位基因对 17 号染色体的生物学效应。最终,它正确预测了这些情况会引发肌萎缩侧索硬化症 (ALS) 。

图示:案例研究示意。(来源:论文)
值得注意的是,BioReason 生成了一个合理的 10 步机制原理,首先识别出 PFN1 基因中特定的 C>G 替换。随后,该模型的推理将该变异与 profilin-1 功能障碍、对细胞骨架完整性至关重要的肌动蛋白动力学受损、随后运动神经元轴突运输中断以及最终 ALS 特有的运动神经元变性联系起来。
结语
总而言之,BioReason 通过将高容量 DNA 序列编码器与大型语言模型的灵活推理能力无缝集成,推动计算生物学的发展,从而构建一个在机制通路推断和变异致病性预测方面均表现卓越的统一框架。DNA-LLM 混合模型的多模态融合,通过强化学习进一步完善,不仅提高了准确性,也为可解释的基因组分析开辟了新的途径。
BioReason:
相关内容:








评论